NNabla Python API デモ・チュートリアル
はじめに、 nnabla およびいくつかの役に立つツールをインポートしましょう。
# python2/3 compatibility
from __future__ import print_function
from __future__ import absolute_import
from __future__ import division
import nnabla as nn # Abbreviate as nn for convenience.
import numpy as np
%matplotlib inline
import matplotlib.pyplot as plt
2017-09-27 14:00:30,785 [nnabla][INFO]: Initializing CPU extension...
NdArray
NdArray is a data container of a multi-dimensional array. NdArray is
device (e.g. CPU, CUDA) and type (e.g. uint8, float32) agnostic, in
which both type and device are implicitly cast or transferred when it
is used. Below, you create a NdArray with a shape of (2, 3, 4).
a = nn.NdArray((2, 3, 4))
以下のように、内部に保持している値を表示することができます。値は初期化されず、デフォルトで float32 として生成されます。
print(a.data)
[[[ 9.42546995e+24 4.56809286e-41 8.47690058e-38 0.00000000e+00]
[ 7.38056336e+34 7.50334969e+28 1.17078231e-32 7.58387310e+31]
[ 7.87001454e-12 9.84394250e-12 6.85712044e+22 1.81785692e+31]]
[[ 1.84681296e+25 1.84933247e+20 4.85656319e+33 2.06176836e-19]
[ 6.80020530e+22 1.69307638e+22 2.11235872e-19 1.94316151e-19]
[ 1.81805047e+31 3.01289097e+29 2.07004908e-19 1.84648795e+25]]]
アクセサである .data は numpy.ndarray として NdArray の値への参照を返します。次のように NumPy API を使うことによって、これらの値を変えることができます。
print('[Substituting random values]')
a.data = np.random.randn(*a.shape)
print(a.data)
print('[Slicing]')
a.data[0, :, ::2] = 0
print(a.data)
[Substituting random values]
[[[ 0.36133638 0.22121875 -1.5912329 -0.33490974]
[ 1.35962474 0.2165522 0.54483992 -0.61813235]
[-0.13718799 -0.44104072 -0.51307833 0.73900551]]
[[-0.59464753 -2.17738533 -0.28626776 -0.45654735]
[ 0.73566747 0.87292582 -0.41605178 0.04792296]
[-0.63856047 0.31966645 -0.63974309 -0.61385244]]]
[Slicing]
[[[ 0. 0.22121875 0. -0.33490974]
[ 0. 0.2165522 0. -0.61813235]
[ 0. -0.44104072 0. 0.73900551]]
[[-0.59464753 -2.17738533 -0.28626776 -0.45654735]
[ 0.73566747 0.87292582 -0.41605178 0.04792296]
[-0.63856047 0.31966645 -0.63974309 -0.61385244]]]
上記の演算はすべてホストデバイス ( CPU ) で行われていることに注意してください。すべての値を定数で埋めたい場合、 NdArray の .zero 、および .fill メソッドを用いてより効率的に行うことができます。データが要求されるとき ( ニューラルネットワークの計算がデータを要求するとき、あるいは、 Python によって NumPy 配列が要求されるとき ) 、それらの値は遅延評価されます。充填演算は特定のデバイス ( 例 CUDA GPU ) 内で実行され、デバイス設定を指定するとより効率的に演算が行われます。これについては後ほど説明します。
a.fill(1) # Filling all values with one.
print(a.data)
[[[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]]
[[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]]]
NumPy 配列オブジェクトから直接 NdArray インスタンスを作ることができます。
b = nn.NdArray.from_numpy_array(np.ones(a.shape))
print(b.data)
[[[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]]
[[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]]]
NdArray は Variable クラスで使用され、ニューラルネットワークの命令型計算でも使用されます。これらについては、後述の章で説明します。
Variable
Variable クラスは、ニューラルネットワークを構築するときに使用されます。ニューラルネットワークは、計算の最小ユニットの演算を定義する Function ( 別名 演算子や層、 Function クラスは後述します ) であるエッジと、 Function の入力 / 出力の値を保持している Variable を表すノードで構成されるグラフとして記述されます。このグラフを “計算グラフ” と呼びます。
NNabla では、計算グラフのノードである Variable は 2 つの NdArray を保持しています。1 つは ( 順方向に計算グラフを実行する ) Forward propagation 中に、 Function の入力値や出力値を格納するためのもので、ニューラルネットワークのパラメータ ( 重み ) へのエラー信号をグラフ上で逆方向に伝播する ) back propagation 中に、エラー信号 ( 勾配 ) を格納するためのものです。 NNabla では、1 つ目を データ と呼び、2 つ目を 勾配 と呼びます。
次の行で、 (2, 3, 4) の形状の Variable インスタンスを作ります。このインスタンスは NdArray として データ と 勾配 を持っています。 need_grad フラグは、False にセットされた場合に backprop 中の不必要な勾配計算を省くために使われます。
x = nn.Variable([2, 3, 4], need_grad=True)
print('x.data:', x.data)
print('x.grad:', x.grad)
x.data: <NdArray((2, 3, 4)) at 0x7f575caf4ea0>
x.grad: <NdArray((2, 3, 4)) at 0x7f575caf4ea0>
以下により、形状を取得することができます。
x.shape
(2, 3, 4)
データ も 勾配 も NdArray なので、 .data アクセサをもつ NdArray のようにその値への参照を取得することができますが、 データ や 勾配 をそれぞれ .d と .g プロパティで参照することもできます。
print('x.data')
print(x.d)
x.d = 1.2345 # To avoid NaN
assert np.all(x.d == x.data.data), 'd: {} != {}'.format(x.d, x.data.data)
print('x.grad')
print(x.g)
x.g = 1.2345 # To avoid NaN
assert np.all(x.g == x.grad.data), 'g: {} != {}'.format(x.g, x.grad.data)
# Zeroing grad values
x.grad.zero()
print('x.grad (after `.zero()`)')
print(x.g)
x.data
[[[ 9.42553452e+24 4.56809286e-41 8.32543479e-38 0.00000000e+00]
[ nan nan 0.00000000e+00 0.00000000e+00]
[ 3.70977305e+25 4.56809286e-41 3.78350585e-44 0.00000000e+00]]
[[ 5.68736600e-38 0.00000000e+00 1.86176378e-13 4.56809286e-41]
[ 4.74367616e+25 4.56809286e-41 5.43829710e+19 4.56809286e-41]
[ 0.00000000e+00 0.00000000e+00 2.93623372e-38 0.00000000e+00]]]
x.grad
[[[ 9.42576510e+24 4.56809286e-41 9.42576510e+24 4.56809286e-41]
[ 9.27127763e-38 0.00000000e+00 9.27127763e-38 0.00000000e+00]
[ 1.69275966e+22 4.80112800e+30 1.21230330e+25 7.22962302e+31]]
[[ 1.10471027e-32 4.63080422e+27 2.44632805e+20 2.87606258e+20]
[ 4.46263300e+30 4.62311881e+30 7.65000750e+28 3.01339003e+29]
[ 2.08627352e-10 1.03961868e+21 7.99576678e+20 1.74441223e+22]]]
x.grad (after .zero())
[[[ 0. 0. 0. 0.]
[ 0. 0. 0. 0.]
[ 0. 0. 0. 0.]]
[[ 0. 0. 0. 0.]
[ 0. 0. 0. 0.]
[ 0. 0. 0. 0.]]]
NdArray のように、 Variable は NumPy 配列から作ることもできます。
x2 = nn.Variable.from_numpy_array(np.ones((3,)), need_grad=True)
print(x2)
print(x2.d)
x3 = nn.Variable.from_numpy_array(np.ones((3,)), np.zeros((3,)), need_grad=True)
print(x3)
print(x3.d)
print(x3.g)
<Variable((3,), need_grad=True) at 0x7f572a5242c8>
[ 1. 1. 1.]
<Variable((3,), need_grad=True) at 0x7f572a5244a8>
[ 1. 1. 1.]
[ 0. 0. 0.]
Variable では、計算グラフ内の値を格納することだけでなく、計算グラフをトレースするために親エッジ ( Function ) を指すことも重要な役割です。ここでは、 x は何の繋がりも持っていません。そのため、 .parent プロパティは None を返します。
print(x.parent)
None
Function
上述のとおり、 Function は計算グラフの演算部分を定義します。モジュール nnabla.functions は様々な Function ( 例 Convolution、 Affine や ReLU ) を提供します。 API リファレンスガイド で利用可能な Function リストを参照してください。
import nnabla.functions as F
例えば、ここでは、入力 Variable に対して要素ごとに Sigmoid Function の出力を計算し、すべての値の和をとる計算グラフを定義するとしましょう。 (これはどのように動作するかを説明するのには十分簡単な例ですが、ニューラルネットワーク学習のコンテキストにおいては意味のない例です。後述でニューラルネットワークの例を示します。)
sigmoid_output = F.sigmoid(x)
sum_output = F.reduce_sum(sigmoid_output)
nnabla.functions では、 Function API は 1 つ ( または複数 ) の Variable と引数 ( あれば ) を取り、 1 つ ( または複数 ) の出力 Variable を返します。 .parent は、それを作成した Function インスタンスを指します。ここではグラフを定義しただけなので、計算は行われないことに注意してください。 ( これは NNabla の計算グラフ API のデフォルトの動作です。グラフ定義中に実際の計算を開始することも可能であり、これは “Dynamic モード” と呼ばれます ( 後ほど説明します ) 。
print("sigmoid_output.parent.name:", sigmoid_output.parent.name)
print("x:", x)
print("sigmoid_output.parent.inputs refers to x:", sigmoid_output.parent.inputs)
sigmoid_output.parent.name: Sigmoid
x: <Variable((2, 3, 4), need_grad=True) at 0x7f572a51a778>
sigmoid_output.parent.inputs refers to x: [<Variable((2, 3, 4), need_grad=True) at 0x7f572a273a48>]
print("sum_output.parent.name:", sum_output.parent.name)
print("sigmoid_output:", sigmoid_output)
print("sum_output.parent.inputs refers to sigmoid_output:", sum_output.parent.inputs)
sum_output.parent.name: ReduceSum
sigmoid_output: <Variable((2, 3, 4), need_grad=True) at 0x7f572a524638>
sum_output.parent.inputs refers to sigmoid_output: [<Variable((2, 3, 4), need_grad=True) at 0x7f572a273a48>]
グラフの末端 Variable で .forward() を呼び出すことで、計算グラフにおける forward propagation を実行します。
sum_output.forward()
print("CG output:", sum_output.d)
print("Reference:", np.sum(1.0 / (1.0 + np.exp(-x.d))))
CG output: 18.59052085876465
Reference: 18.5905
.backward() は、グラフ乗で back propagation を行います。 NNabla の backprop アルゴリズムでは、入力 Variable に対して勾配を積算しているので、ここでは、backprop の前に 勾配 の値を 0 に初期化します。
x.grad.zero()
sum_output.backward()
print("d sum_o / d sigmoid_o:")
print(sigmoid_output.g)
print("d sum_o / d x:")
print(x.g)
d sum_o / d sigmoid_o:
[[[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]]
[[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]
[ 1. 1. 1. 1.]]]
d sum_o / d x:
[[[ 0.17459197 0.17459197 0.17459197 0.17459197]
[ 0.17459197 0.17459197 0.17459197 0.17459197]
[ 0.17459197 0.17459197 0.17459197 0.17459197]]
[[ 0.17459197 0.17459197 0.17459197 0.17459197]
[ 0.17459197 0.17459197 0.17459197 0.17459197]
[ 0.17459197 0.17459197 0.17459197 0.17459197]]]
NNabla は主にニューラルネットワークの学習と推論に焦点を当てて開発されています。ニューラルネットワークには、 Convolution、 Affine ( 別名 全結合、 Dense 、など ) のように、計算ブロックと付随して学習可能なパラメータがあります。 NNabla では、学習可能なパラメータも Variable オブジェクトとして表されています。入力 Variable と同様に、それらのパラメータ Variable も Function に渡すことによって使われます。例えば、 Affine 関数は入力、重み、そしてバイアスを入力として受け取ります。
x = nn.Variable([5, 2]) # Input
w = nn.Variable([2, 3], need_grad=True) # Weights
b = nn.Variable([3], need_grad=True) # Biases
affine_out = F.affine(x, w, b) # Create a graph including only affine
上記の例は、 B = 5 ( バッチサイズ ) および D = 2 ( 次元 ) の入力を受け取り、それを D’ = 3 出力、すなわち (B , D’) 出力にマップします。
ここで、パラメータ Variable ( w と b ) に対してだけ need_grad=True をセットしました。 x は非パラメータ Variable で、計算グラフのルートです。従って、 x は勾配計算が必要ありません。これにより、 x に対する勾配計算は最初のaffineでは実行されず、不必要なバックプロパゲーションの計算を省くことができます。
次の部分で、データをセットし、勾配を初期化し、それから順方向の計算と逆方向の計算を行います。
# Set random input and parameters
x.d = np.random.randn(*x.shape)
w.d = np.random.randn(*w.shape)
b.d = np.random.randn(*b.shape)
# Initialize grad
x.grad.zero() # Just for showing gradients are not computed when need_grad=False (default).
w.grad.zero()
b.grad.zero()
# Forward and backward
affine_out.forward()
affine_out.backward()
# Note: Calling backward at non-scalar Variable propagates 1 as error message from all element of outputs. .
affine_out が Affine の出力を保持します。
print('F.affine')
print(affine_out.d)
print('Reference')
print(np.dot(x.d, w.d) + b.d)
F.affine
[[-0.17701732 2.86095762 -0.82298267]
[-0.75544345 -1.16702223 -2.44841242]
[-0.36278027 -3.4771595 -0.75681627]
[ 0.32743117 0.24258983 1.30944324]
[-0.87201929 1.94556415 -3.23357344]]
Reference
[[-0.1770173 2.86095762 -0.82298267]
[-0.75544345 -1.16702223 -2.44841242]
[-0.3627803 -3.4771595 -0.75681627]
[ 0.32743117 0.24258983 1.309443 ]
[-0.87201929 1.94556415 -3.23357344]]
結果として重みとバイアスの勾配は次のようになります。
print("dw")
print(w.g)
print("db")
print(b.g)
dw
[[ 3.10820675 3.10820675 3.10820675]
[ 0.37446201 0.37446201 0.37446201]]
db
[ 5. 5. 5.]
need_grad が False にセットされているため、 x の勾配は変わりません。
print(x.g)
[[ 0. 0.]
[ 0. 0.]
[ 0. 0.]
[ 0. 0.]
[ 0. 0.]]
Parametric function
Function の入力としてのパラメータを考えることにより、計算グラフの表現性と柔軟性が高まります。しかし、学習できる Function それぞれに対してすべてのパラメータを定義することは、ニューラルネットワークを定義するユーザーにとって面倒です。 NNabla では、学習できるモデルは通常、最適化可能なパラメータをもつ Function を構成することによって作られます。 これらの Function は "Parametric function" と呼ばれています。 Parametric function API は、様々な Parametric function と学習できるモデルを構成するためのインターフェイスを提供します。
Parametric function を使うために、以下のようにインポートを行います。
import nnabla.parametric_functions as PF
最適化可能なパラメータをもつ Function は以下のように作ることができます。
with nn.parameter_scope("affine1"):
c1 = PF.affine(x, 3)
1 行目で parameter scope を作っています。そして、 2 行目で PF.affine - affine変換 - を x に対して適用し、その結果を保持する変数 c1 を作っています。パラメータは関数呼び出しで作られ、ランダムに初期化され、 parameter_scope コンテキストを使って “affine1” という名前で登録されます。関数 nnabla.get_parameters() で登録されたパラメータを取得することができます。
nn.get_parameters()
OrderedDict([('affine1/affine/W',
<Variable((2, 3), need_grad=True) at 0x7f572822f0e8>),
('affine1/affine/b',
<Variable((3,), need_grad=True) at 0x7f572822f138>)])
The name= argument of any PF function creates the equivalent
parameter space to the above definition of PF.affine transformation
as below. It could save the space of your Python code. The
nnabla.parameter_scope is more useful when you group multiple
parametric functions such as Convolution-BatchNormalization found in a
typical unit of CNNs.
c1 = PF.affine(x, 3, name='affine1')
nn.get_parameters()
OrderedDict([('affine1/affine/W',
<Variable((2, 3), need_grad=True) at 0x7f572822f0e8>),
('affine1/affine/b',
<Variable((3,), need_grad=True) at 0x7f572822f138>)])
出力およびパラメータの形状 ( 上記のとおり ) が affine 変換の出力サイズ ( 上記の例では出力サイズは 3 ) により自動的に決まるということは注目すべき点です。これにより、簡単にグラフを作ることができます。
c1.shape
(5, 3)
( 特に意味のない例ですが ) パラメータのスコープは次のように入れ子にすることができます。
with nn.parameter_scope('foo'):
h = PF.affine(x, 3)
with nn.parameter_scope('bar'):
h = PF.affine(h, 4)
結果として以下が作成されます。
nn.get_parameters()
OrderedDict([('affine1/affine/W',
<Variable((2, 3), need_grad=True) at 0x7f572822f0e8>),
('affine1/affine/b',
<Variable((3,), need_grad=True) at 0x7f572822f138>),
('foo/affine/W',
<Variable((2, 3), need_grad=True) at 0x7f572822fa98>),
('foo/affine/b',
<Variable((3,), need_grad=True) at 0x7f572822fae8>),
('foo/bar/affine/W',
<Variable((3, 4), need_grad=True) at 0x7f572822f728>),
('foo/bar/affine/b',
<Variable((4,), need_grad=True) at 0x7f572822fdb8>)])
また、 get_parameters() は parameter_scope で使用できます。例えば、
with nn.parameter_scope("foo"):
print(nn.get_parameters())
OrderedDict([('affine/W', <Variable((2, 3), need_grad=True) at 0x7f572822fa98>), ('affine/b', <Variable((3,), need_grad=True) at 0x7f572822fae8>), ('bar/affine/W', <Variable((3, 4), need_grad=True) at 0x7f572822f728>), ('bar/affine/b', <Variable((4,), need_grad=True) at 0x7f572822fdb8>)])
nnabla.clear_parameters() により、スコープの中で登録されたパラメータを消去することができます。
with nn.parameter_scope("foo"):
nn.clear_parameters()
print(nn.get_parameters())
OrderedDict([('affine1/affine/W', <Variable((2, 3), need_grad=True) at 0x7f572822f0e8>), ('affine1/affine/b', <Variable((3,), need_grad=True) at 0x7f572822f138>)])
多層パーセプトロンのサンプル
次のブロックは、 2 層の完全結合のニューラルネットワーク ( 多層パーセプトロン ) によって、2 次元の入力から 1 次元の出力を予測するための計算グラフを作ります。
nn.clear_parameters()
batchsize = 16
x = nn.Variable([batchsize, 2])
with nn.parameter_scope("fc1"):
h = F.tanh(PF.affine(x, 512))
with nn.parameter_scope("fc2"):
y = PF.affine(h, 1)
print("Shapes:", h.shape, y.shape)
Shapes: (16, 512) (16, 1)
結果として次のようなパラメータ Variable が作成されます。
nn.get_parameters()
OrderedDict([('fc1/affine/W',
<Variable((2, 512), need_grad=True) at 0x7f572822fef8>),
('fc1/affine/b',
<Variable((512,), need_grad=True) at 0x7f572822f9a8>),
('fc2/affine/W',
<Variable((512, 1), need_grad=True) at 0x7f572822f778>),
('fc2/affine/b',
<Variable((1,), need_grad=True) at 0x7f572822ff98>)])
上記で説明したように、末端の Variable で順方向メソッドを呼び出すことで順方向パスを実行することができます。
x.d = np.random.randn(*x.shape) # Set random input
y.forward()
print(y.d)
[[-0.05708594]
[ 0.01661986]
[-0.34168088]
[ 0.05822293]
[-0.16566885]
[-0.04867431]
[ 0.2633169 ]
[ 0.10496549]
[-0.01291842]
[-0.09726256]
[-0.05720493]
[-0.09691752]
[-0.07822668]
[-0.17180404]
[ 0.11970415]
[-0.08222144]]
ニューラルネットワークの学習では、 backprop を用いた勾配降下法によって loss の値を最小化する必要があります。 NNabla では、 loss 関数は単一の Function であり、 Function モジュールに含まれます。
# Variable for label
label = nn.Variable([batchsize, 1])
# Set loss
loss = F.reduce_mean(F.squared_error(y, label))
# Execute forward pass.
label.d = np.random.randn(*label.shape) # Randomly generate labels
loss.forward()
print(loss.d)
1.9382084608078003
上記のとおり、 NNabla で backward を実行するとルートの Variable で勾配を蓄積します。そのため、 backprop の前にパラメータ Variable の勾配を初期化する必要があります ( 別途 Solver APIで簡単な方法を説明します ) 。
# Collect all parameter variables and init grad.
for name, param in nn.get_parameters().items():
param.grad.zero()
# Gradients are accumulated to grad of params.
loss.backward()
Imperative モード
backprop を実行すると、勾配はパラメータ Variable の grad 領域に保持されます。次のブロックでは、基本的な勾配降下法でパラメータを更新します。
for name, param in nn.get_parameters().items():
param.data -= param.grad * 0.001 # 0.001 as learning rate
上記の計算は、ニューラルネットワークを実行するための NNabla の “ Imperative モード” の例です。通常、 NNabla Function ( nnabla.functions のインスタンス ) は入力として Variable を取ります。 ( Variable の代わりに ) NNabla Function への入力として少なくとも 1 つの NdArray が与えられると、 Function の計算はすぐに開始し、出力として Variable を返す代わりに NdArray を返します。上記の例で、NNabla Function F.mul_scalar と F.sub2 がそれぞれオーバーライドされた演算子 * と -= によって呼び出されます。
つまり、 NNabla の “ Imperative モード ” は計算グラフを作成しませんが、NumPy のように使うことができます。もし CUDA のようなデバイスアクセラレーションが有効ならば、それにより高速化された NumPy のように使うことができます。 Parametric function は NdArray 入力でも使うことができます。次のブロックでは、簡単な Imperative 実行の例を説明します。
# A simple example of imperative mode.
xi = nn.NdArray.from_numpy_array(np.arange(4).reshape(2, 2))
yi = F.relu(xi - 1)
print(xi.data)
print(yi.data)
[[0 1]
[2 3]]
[[ 0. 0.]
[ 1. 2.]]
右辺から左辺への in-place な代入は、 = 演算子では適切に行えないことに注意してください。 例えば、 x が NdArray であるとき、 x = x + 1 により x の値が増加するわけでは ありません。 その代わりに、 x がもともと参照されていた NdArray とは異なる、右辺で新たに作成された NdArray を左辺の x は参照します。
For in-place editing of NdArrays, the in-place assignment operators
+=, -=, *=, and /= can be used. The copy_from method
can also be used to copy values of an existing NdArray to another.
For example, increment 1 to x, an NdArray, can be done by
x.copy_from(x+1). The copy is performed with device acceleration if
a device context is specified by using nnabla.set_default_context or
nnabla.context_scope.
# The following doesn't perform substitution but assigns a new NdArray object to `xi`.
# xi = xi + 1
# The following copies the result of `xi + 1` to `xi`.
xi.copy_from(xi + 1)
assert np.all(xi.data == (np.arange(4).reshape(2, 2) + 1))
# Inplace operations like `+=`, `*=` can also be used (more efficient).
xi += 1
assert np.all(xi.data == (np.arange(4).reshape(2, 2) + 2))
Solver
NNabla は nnabla.solvers モジュールでは、パラメータを最適化するために確率的勾配降下法アルゴリズムを提供します。上記で説明したパラメータの更新はこの Solver API で置き換えることができ、この API はより簡単であり、大抵の場合より速いです。
from nnabla import solvers as S
solver = S.Sgd(lr=0.00001)
solver.set_parameters(nn.get_parameters())
# Set random data
x.d = np.random.randn(*x.shape)
label.d = np.random.randn(*label.shape)
# Forward
loss.forward()
以下の solver メソッドを呼び出し、勾配領域を 0 で埋めたあと、 backprop を呼び出します。
solver.zero_grad()
loss.backward()
次のブロックでは、通常の SGD 更新則を使ってパラメータを更新します (上記の Imperative の例と同様 ) 。
solver.update()
トイ問題を利用した学習の例
以下の関数のようなベクトルのノルムの計算を回帰する問題を考えます。
def vector2length(x):
# x : [B, 2] where B is number of samples.
return np.sqrt(np.sum(x ** 2, axis=1, keepdims=True))
次のように、 matplotlib の contour plot により、この変換をビジュアライズします。
# Data for plotting contour on a grid data.
xs = np.linspace(-1, 1, 100)
ys = np.linspace(-1, 1, 100)
grid = np.meshgrid(xs, ys)
X = grid[0].flatten()
Y = grid[1].flatten()
def plot_true():
"""Plotting contour of true mapping from a grid data created above."""
plt.contourf(xs, ys, vector2length(np.hstack([X[:, None], Y[:, None]])).reshape(100, 100))
plt.axis('equal')
plt.colorbar()
plot_true()

これらを予測する深層ニューラルネットワークを定義します。
def length_mlp(x):
h = x
for i, hnum in enumerate([4, 8, 4, 2]):
h = F.tanh(PF.affine(h, hnum, name="fc{}".format(i)))
y = PF.affine(h, 1, name='fc')
return y
nn.clear_parameters()
batchsize = 100
x = nn.Variable([batchsize, 2])
y = length_mlp(x)
label = nn.Variable([batchsize, 1])
loss = F.reduce_mean(F.squared_error(y, label))
for ループを使って 5 層の深層 MLP を作成しました。わずか 3 行のコードだけで無限に深いニューラルネットワークを作成可能です。次のブロックでは、学習済み関数を視覚化するための helper 関数を追加します。
def predict(inp):
ret = []
for i in range(0, inp.shape[0], x.shape[0]):
xx = inp[i:i + x.shape[0]]
# Imperative execution
xi = nn.NdArray.from_numpy_array(xx)
yi = length_mlp(xi)
ret.append(yi.data.copy())
return np.vstack(ret)
def plot_prediction():
plt.contourf(xs, ys, predict(np.hstack([X[:, None], Y[:, None]])).reshape(100, 100))
plt.colorbar()
plt.axis('equal')
続いて、 solver オブジェクトを作成します。この分野でもっともよく使用される SGD アルゴリズムの 1 つである Adam オプティマイザーを使います。
from nnabla import solvers as S
solver = S.Adam(alpha=0.01)
solver.set_parameters(nn.get_parameters())
次の関数は真のシステムにより無限にデータを生成します。
def random_data_provider(n):
x = np.random.uniform(-1, 1, size=(n, 2))
y = vector2length(x)
return x, y
次のブロックでは、 2000 回の学習ステップ ( SGD による更新 ) を実行します。
num_iter = 2000
for i in range(num_iter):
# Sample data and set them to input variables of training.
xx, ll = random_data_provider(batchsize)
x.d = xx
label.d = ll
# Forward propagation given inputs.
loss.forward(clear_no_need_grad=True)
# Parameter gradients initialization and gradients computation by backprop.
solver.zero_grad()
loss.backward(clear_buffer=True)
# Apply weight decay and update by Adam rule.
solver.weight_decay(1e-6)
solver.update()
# Just print progress.
if i % 100 == 0 or i == num_iter - 1:
print("Loss@{:4d}: {}".format(i, loss.d))
Loss@ 0: 0.6976373195648193
Loss@ 100: 0.08075223118066788
Loss@ 200: 0.005213144235312939
Loss@ 300: 0.001955194864422083
Loss@ 400: 0.0011660841992124915
Loss@ 500: 0.0006421314901672304
Loss@ 600: 0.0009330055327154696
Loss@ 700: 0.0008817618945613503
Loss@ 800: 0.0006205961108207703
Loss@ 900: 0.0009072928223758936
Loss@1000: 0.0008160348515957594
Loss@1100: 0.0011569359339773655
Loss@1200: 0.000837412488181144
Loss@1300: 0.0011542742140591145
Loss@1400: 0.0005833200993947685
Loss@1500: 0.0009848927147686481
Loss@1600: 0.0005141657311469316
Loss@1700: 0.0009339841199107468
Loss@1800: 0.000950580753851682
Loss@1900: 0.0005430278833955526
Loss@1999: 0.0007046313839964569
Memory usage optimization: You may notice that, in the above
updates, .forward() is called with the clear_no_need_grad=
option, and .backward() is called with the clear_buffer= option.
Training of neural network in more realistic scenarios usually consumes
huge memory due to the nature of backpropagation algorithm, in which all
of the forward variable buffer data should be kept in order to
compute the gradient of a function. In a naive implementation, we keep
all the variable data and grad living until the NdArray
objects are not referenced (i.e. the graph is deleted). The clear_*
options in .forward() and .backward() enables to save memory
consumption due to that by clearing (erasing) memory of data and
grad when it is not referenced by any subsequent computation. (More
precisely speaking, it doesn't free memory actually. We use our memory
pool engine by default to avoid memory alloc/free overhead). The
unused buffers can be re-used in subsequent computation. See the
document of Variable for more details. Note that the following
loss.forward(clear_buffer=True) clears data of any intermediate
variables. If you are interested in intermediate variables for some
purposes (e.g. debug, log), you can use the .persistent flag to
prevent clearing buffer of a specific Variable like below.
loss.forward(clear_buffer=True)
print("The prediction `y` is cleared because it's an intermediate variable.")
print(y.d.flatten()[:4]) # to save space show only 4 values
y.persistent = True
loss.forward(clear_buffer=True)
print("The prediction `y` is kept by the persistent flag.")
print(y.d.flatten()[:4]) # to save space show only 4 value
The predictionyis cleared because it's an intermediate variable. [ 2.27279830e-04 6.02164946e-05 5.33679675e-04 2.35557582e-05] The predictionyis kept by the persistent flag. [ 1.0851264 0.87657517 0.79603785 0.40098712]
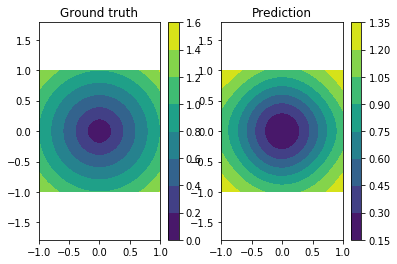
次のように正解と予測関数の視覚化により、予測がかなりうまく行われていることを確認することができました。
plt.subplot(121)
plt.title("Ground truth")
plot_true()
plt.subplot(122)
plt.title("Prediction")
plot_prediction()
nnabla.save_parameters によって学習されたパラメータを保存し、 nnabla.load_parameters によって読み込むことができます。
path_param = "param-vector2length.h5"
nn.save_parameters(path_param)
# Remove all once
nn.clear_parameters()
nn.get_parameters()
2017-09-27 14:00:40,544 [nnabla][INFO]: Parameter save (.h5): param-vector2length.h5
OrderedDict()
# Load again
nn.load_parameters(path_param)
print('\n'.join(map(str, nn.get_parameters().items())))
2017-09-27 14:00:40,564 [nnabla][INFO]: Parameter load (<built-in function format>): param-vector2length.h5
('fc0/affine/W', <Variable((2, 4), need_grad=True) at 0x7f576328df48>)
('fc0/affine/b', <Variable((4,), need_grad=True) at 0x7f57245f2868>)
('fc1/affine/W', <Variable((4, 8), need_grad=True) at 0x7f576328def8>)
('fc1/affine/b', <Variable((8,), need_grad=True) at 0x7f5727ee5c78>)
('fc2/affine/W', <Variable((8, 4), need_grad=True) at 0x7f5763297318>)
('fc2/affine/b', <Variable((4,), need_grad=True) at 0x7f5727d29908>)
('fc3/affine/W', <Variable((4, 2), need_grad=True) at 0x7f57632973b8>)
('fc3/affine/b', <Variable((2,), need_grad=True) at 0x7f57632974a8>)
('fc/affine/W', <Variable((2, 1), need_grad=True) at 0x7f57632974f8>)
('fc/affine/b', <Variable((1,), need_grad=True) at 0x7f5763297598>)
save および load 関数はパラメータスコープ内で使用できます。
with nn.parameter_scope('foo'):
nn.load_parameters(path_param)
print('\n'.join(map(str, nn.get_parameters().items())))
2017-09-27 14:00:40,714 [nnabla][INFO]: Parameter load (<built-in function format>): param-vector2length.h5
('fc0/affine/W', <Variable((2, 4), need_grad=True) at 0x7f576328df48>)
('fc0/affine/b', <Variable((4,), need_grad=True) at 0x7f57245f2868>)
('fc1/affine/W', <Variable((4, 8), need_grad=True) at 0x7f576328def8>)
('fc1/affine/b', <Variable((8,), need_grad=True) at 0x7f5727ee5c78>)
('fc2/affine/W', <Variable((8, 4), need_grad=True) at 0x7f5763297318>)
('fc2/affine/b', <Variable((4,), need_grad=True) at 0x7f5727d29908>)
('fc3/affine/W', <Variable((4, 2), need_grad=True) at 0x7f57632973b8>)
('fc3/affine/b', <Variable((2,), need_grad=True) at 0x7f57632974a8>)
('fc/affine/W', <Variable((2, 1), need_grad=True) at 0x7f57632974f8>)
('fc/affine/b', <Variable((1,), need_grad=True) at 0x7f5763297598>)
('foo/fc0/affine/W', <Variable((2, 4), need_grad=True) at 0x7f5763297958>)
('foo/fc0/affine/b', <Variable((4,), need_grad=True) at 0x7f57632978b8>)
('foo/fc1/affine/W', <Variable((4, 8), need_grad=True) at 0x7f572a51ac78>)
('foo/fc1/affine/b', <Variable((8,), need_grad=True) at 0x7f5763297c78>)
('foo/fc2/affine/W', <Variable((8, 4), need_grad=True) at 0x7f5763297a98>)
('foo/fc2/affine/b', <Variable((4,), need_grad=True) at 0x7f5763297d68>)
('foo/fc3/affine/W', <Variable((4, 2), need_grad=True) at 0x7f5763297e08>)
('foo/fc3/affine/b', <Variable((2,), need_grad=True) at 0x7f5763297ea8>)
('foo/fc/affine/W', <Variable((2, 1), need_grad=True) at 0x7f5763297f48>)
('foo/fc/affine/b', <Variable((1,), need_grad=True) at 0x7f5763297cc8>)
!rm {path_param} # Clean ups