固定小数点量子化によるネットワークの圧縮¶
このチュートリアルでは、固定小数点量子化を利用してネットワークを圧縮する方法を紹介します。
はじめに¶
ニューラルネットワークは、物体認識や検出などの AI フィールドで信頼できる結果をもたらし、実際のアプリケーションで役立ちます。認識技術の進歩と共に、これらの技術を利用したIoTエッジデバイスが増加しており、データセンターでは膨大なデータ量を処理する必要があります。その結果、デバイスとデータセンターのネットワークの帯域幅の要件は限界まで引き上げられています。ネットワークテクノロジーが改善される一方、データセンターは、多くのアプリケーションにとって重要な要件となりえる許容可能な転送速度や応答時間を保証することができません。そこで、データセンターで処理するのではなく、IoTデバイス上で認識を実行することを考えます。しかし、CNN ベースの認識システムは大量のメモリと計算能力が必要であり、それらは高価な GPU をベースとしたマシン上で高い性能を発揮します。例えば、 AlexNet には 61M のパラメータ( 249MB のメモリ)があり、1.5B の高精度演算を実行して 1 つの画像を分類します。これらの数値は、VGG などのより複雑な CNN の場合はさらに大きくなります。これらは多くの場合、携帯電話、小型デバイス、組み込み電子機器などのエッジデバイスには適さないでしょう。したがって、ネットワーク容量と計算の複雑さを減らすことがモデルの最適化として求められています。
現在、元の精度を保ったまま、メモリと計算時間を大幅に削減する方法として、重みと(または)入力の量子化が知られています [1] (*) 。
このチュートリアルでは、nnabla を利用したこれらの最適化の実施例をご紹介します。
2つのアプローチ¶
固定小数点を利用した深層畳み込みネットワークを設計する方法として、主に2つのアプローチがあります。
固定小数点制約でネットワークを学習する
浮動小数点演算を利用して学習したネットワークを、固定小数点演算で実行できるように変換する
BinaryConnectAffine, BinaryConnectConvolution などの Binary Connect をサポートするような関数と、 BinaryWeightAffine, BinaryWeightConvolution などの Binary Weight をサポートするような関数、FixedPointQuantizedAffine, FixedPointQuantizedConvolution などの固定小数点量子化をサポートするような関数は、最初のアプローチで重要な役割を果たします。これらの関数には、forward演算とbackward演算で異なるデータパスがあります。forward演算実行時は、浮動小数点の重みが固定小数点の重みまたはバイナリの重みに変換され、出力は、入力と、これらの二値化された(または量子化された)重みによって計算されます。backward演算実行時は、浮動小数点の(学習可能な)重みのみが更新に利用されます。実際のところ、重みの 2 値化は極端すぎるため、固定小数点による量子化がより一般的に使用されているようです。。
2 番目のアプローチにより、精度の低下を最小限に抑えるために、いくつかの手順を実行する必要があります。[2] では、量子化サイズとパフォーマンスの関係について、包括的に分析しています。ここでは、精度とストレージサイズ間でのトレードオフを示すために、いくつかの実験を行います。
ResNet23 の元のバージョンと binary_weight_convolution() を使用したバージョンを比較したところ、binary_weight_convolution() を使用した後の精度は [3] で報告されている精度よりも劣化しています。この結果は、より慎重にファインチューニングすることで、改善される可能性があります。
このチュートリアルでは、ネットワークを圧縮する基本的な方法のみを示します。このチュートリアルで紹介されていない新しいアプローチはまだまだたくさんあります。例えば、 [4] で提案された新しい手法では、量子化表現のネットワークを生徒、浮動小数点演算による高精度なネットワークを教師として、ネットワークの蒸留により学習を行うことで、最先端のパフォーマンスと精度を達成しています。
ベンチマーク¶
ここでは、べンチマークとして、シンプルなネットワークの ResNet23 および CIFAR-10 データセットを選択しました。まず、精度、モデルサイズなど、このベンチマークの基本メトリックを取得します。以下にネットワーク構造を示します。
層の名前 |
形状 |
必要なバッファサイズ |
|---|---|---|
conv1 |
(1, 3, 32, 32) -> (1, 64, 32, 32) |
281344 |
conv2 |
(1, 64, 32, 32)->(1, 32, 32, 32)->(1, 32, 32, 32) -> (1, 64, 32, 32) |
786432 |
conv3 |
(1, 64, 16, 16)->(1, 32, 16, 16)->(1, 32, 16, 16) -> (1, 64, 16, 16) |
196608 |
conv4 |
(1, 64, 16, 16)->(1, 32, 16, 16)->(1, 32, 16, 16) -> (1, 64, 16, 16) |
196608 |
conv5 |
(1, 64, 8, 8)->(1, 32, 8, 8)->(1, 32, 8, 8) -> (1, 64, 8, 8) |
49152 |
conv6 |
(1, 64, 8, 8)->(1, 32, 8, 8)->(1, 32, 8, 8) -> (1, 64, 8, 8) |
49152 |
conv7 |
(1, 64, 4, 4)->(1, 32, 4, 4)->(1, 32, 4, 4) -> (1, 64, 4, 4) |
12288 |
conv8 |
(1, 64, 4, 4)->(1, 32, 4, 4)->(1, 32, 4, 4) -> (1, 64, 4, 4) |
12288 |
フットプリントを小さくするためには、パラメータのサイズとVariableのバッファーサイズについて考える必要があります。特に、最大バッファーサイズを優先的に検討する必要があるでしょう。
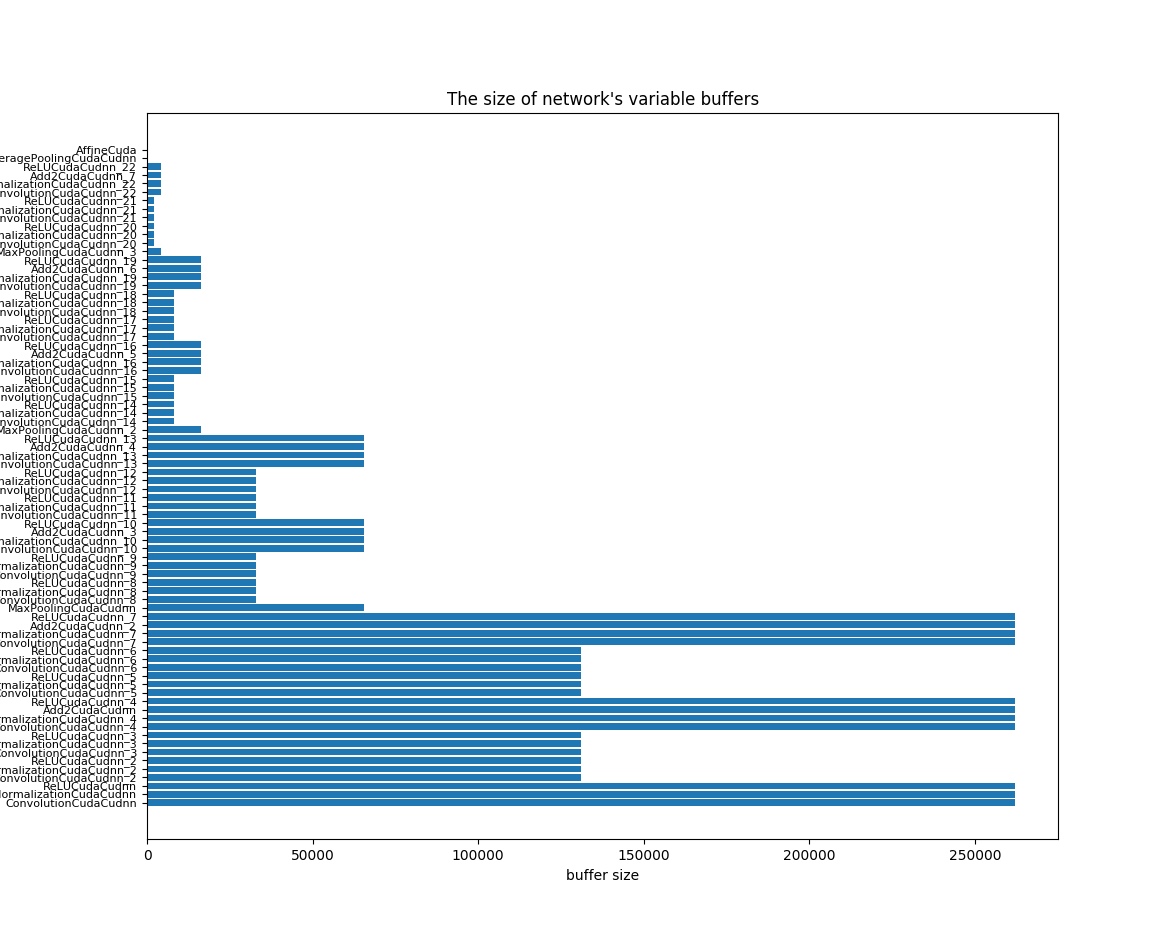
ネットワークは、次のコードによって作成されます。
import nnabla as nn
import nnabla.functions as F
import nnabla.parametric_functions as PF
def resnet23_prediction(image, test=False, ncls=10, nmaps=64, act=F.relu):
"""
Construct ResNet 23
"""
# Residual Unit
def res_unit(x, scope_name, dn=False):
C = x.shape[1]
with nn.parameter_scope(scope_name):
# Conv -> BN -> Nonlinear
with nn.parameter_scope("conv1"):
h = PF.convolution(x, C // 2, kernel=(1, 1), pad=(0, 0),
with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
h = act(h)
# Conv -> BN -> Nonlinear
with nn.parameter_scope("conv2"):
h = PF.convolution(h, C // 2, kernel=(3, 3), pad=(1, 1),
with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
h = act(h)
# Conv -> BN
with nn.parameter_scope("conv3"):
h = PF.convolution(h, C, kernel=(1, 1), pad=(0, 0),
with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
# Residual -> Nonlinear
h = act(F.add2(h, x, inplace=True))
# Maxpooling
if dn:
h = F.max_pooling(h, kernel=(2, 2), stride=(2, 2))
return h
# Conv -> BN -> Nonlinear
with nn.parameter_scope("conv1"):
# Preprocess
if not test:
image = F.image_augmentation(image, contrast=1.0,
angle=0.25,
flip_lr=True)
image.need_grad = False
h = PF.convolution(image, nmaps, kernel=(3, 3),
pad=(1, 1), with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
h = act(h)
h = res_unit(h, "conv2", False) # -> 32x32
h = res_unit(h, "conv3", True) # -> 16x16
h = res_unit(h, "conv4", False) # -> 16x16
h = res_unit(h, "conv5", True) # -> 8x8
h = res_unit(h, "conv6", False) # -> 8x8
h = res_unit(h, "conv7", True) # -> 4x4
h = res_unit(h, "conv8", False) # -> 4x4
h = F.average_pooling(h, kernel=(4, 4)) # -> 1x1
pred = PF.affine(h, ncls)
return pred
次の図のように、トップ 1 エラーは 0.16 に達します。
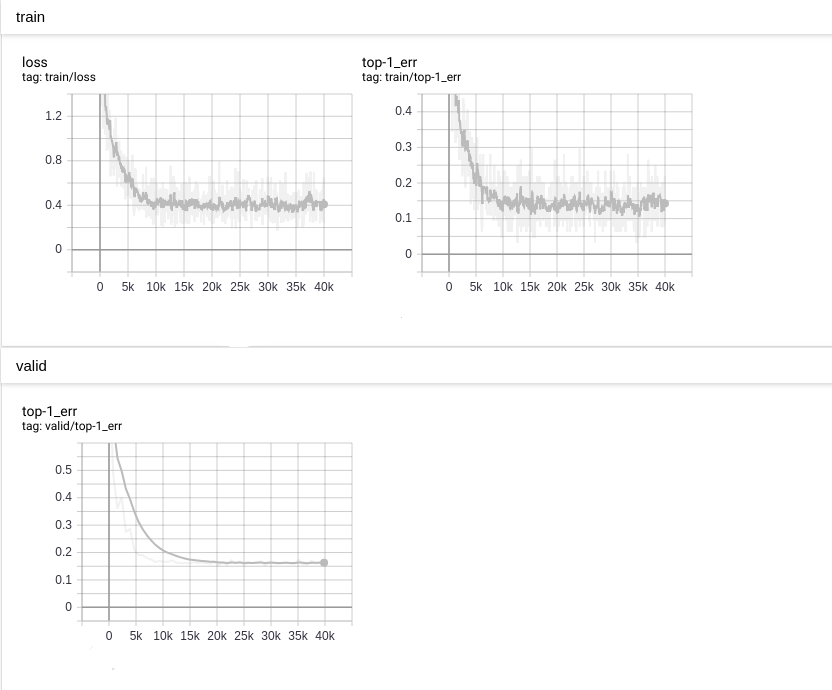
CIFAR10 テストデータセットで、 nnabla_cli infer と nnablart infer 間の精度を比較しました。比較コードは以下の通りです。
import numpy as np
import os
from cifar10_data import data_iterator_cifar10
data_iterator = data_iterator_cifar10
vdata = data_iterator(1, False)
iter_num = 100
def get_infer_result(result_file):
d0 = np.fromfile(result_file, np.float32)
d0 = d0.reshape((10, ))
return np.argmax(d0)
def normalize_image(image):
image = image.astype(np.float32)
image -= np.mean(image)
image_std = np.std(image)
return image / max(image_std, 1e-5)
nnp_correct = 0
nnb_correct = 0
for i in range(iter_num):
img, gt = vdata.next()
img = normalize_image(img)
img.tofile('input.bin')
os.system('nnabla_cli infer -b 1 -c bin_class.nnp -o output_0 input.bin')
os.system('./nnablart infer bin_class.nnb input.bin output_1')
r1 = get_infer_result('output_0_0.bin')
r2 = get_infer_result('output_1_0.bin')
if r1 == gt:
nnp_correct += 1
if r2 == gt:
nnb_correct += 1
if r1 == r2 == gt:
print("{}: all same!".format(i))
else:
print("{}: not all same".format(i))
print("nnp accuracy: {}, nnb accuracy: {}".format(
float(nnp_correct) / iter_num, float(nnb_correct) / iter_num))
このコードでは、 nnablart は nnabla-c-runtime に基づいて実装された実行可能ファイルです。 nnablart は、 *.nnb ファイルで定義されたネットワークを推測できるシンプルなコマンドラインインターフェイスです。ご存知のように、 nnabla-c-runtime は、利用できるメモリに制約のある小さなデバイス上での実行を目的としてc言語で実装されています。メモリポリシーを慎重に設計し、またメモリ量削減のために学習に利用するコードを削除しています。このテストプログラムは、100 サンプルに対して反復実行され、Ground Truthと比較して、精度を計算します。
...
NNabla command line interface (Version:1.0.18, Build:190619071959)
0: input.bin
1: output_1
Input[0] size:3072
Input[0] data type:NN_DATA_TYPE_FLOAT, fp:0
Input[0] Shape ( 1 3 32 32 )
Output[0] size:10
Output[0] filename output_1_0.bin
Output[0] Shape ( 1 10 )
Output[0] data type:NN_DATA_TYPE_FLOAT, fp:0
99: all same!
nnp accuracy: 0.81, nnb accuracy: 0.81
binary_weight_convolution¶
次のように、 PF.convolution() を PF.binary_weight_convolution() に置き換えた例を示します。
import nnabla as nn
import nnabla.functions as F
import nnabla.parametric_functions as PF
def resnet23_bin_w(image, test=False, ncls=10, nmaps=64, act=F.relu):
"""
Construct ResNet 23
"""
# Residual Unit
def res_unit(x, scope_name, dn=False):
C = x.shape[1]
with nn.parameter_scope(scope_name):
# Conv -> BN -> Nonlinear
with nn.parameter_scope("conv1"):
h = PF.binary_weight_convolution(x, C // 2, kernel=(1, 1), pad=(0, 0),
with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
h = act(h)
# Conv -> BN -> Nonlinear
with nn.parameter_scope("conv2"):
h = PF.binary_weight_convolution(h, C // 2, kernel=(3, 3), pad=(1, 1),
with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
h = act(h)
# Conv -> BN
with nn.parameter_scope("conv3"):
h = PF.binary_weight_convolution(h, C, kernel=(1, 1), pad=(0, 0),
with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
# Residual -> Nonlinear
h = act(F.add2(h, x, inplace=True))
# Maxpooling
if dn:
h = F.max_pooling(h, kernel=(2, 2), stride=(2, 2))
return h
# Conv -> BN -> Nonlinear
with nn.parameter_scope("conv1"):
# Preprocess
if not test:
image = F.image_augmentation(image, contrast=1.0,
angle=0.25,
flip_lr=True)
image.need_grad = False
h = PF.binary_weight_convolution(image, nmaps, kernel=(3, 3),
pad=(1, 1), with_bias=False)
h = PF.batch_normalization(h, batch_stat=not test)
h = act(h)
h = res_unit(h, "conv2", False) # -> 32x32
h = res_unit(h, "conv3", True) # -> 16x16
h = res_unit(h, "conv4", False) # -> 16x16
h = res_unit(h, "conv5", True) # -> 8x8
h = res_unit(h, "conv6", False) # -> 8x8
h = res_unit(h, "conv7", True) # -> 4x4
h = res_unit(h, "conv8", False) # -> 4x4
h = F.average_pooling(h, kernel=(4, 4)) # -> 1x1
pred = PF.affine(h, ncls)
return pred
以下のような学習速度の劣化と精度劣化が観測されました。
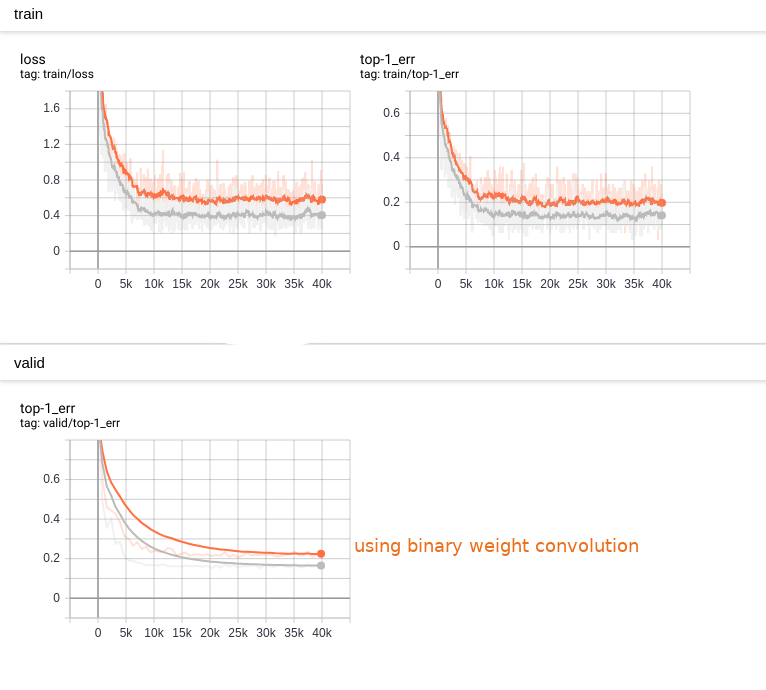
モデルとパラメータを *.nnp ファイルとして保存しました。次に、メモリ制約デバイスに適合するように、 *.nnb に変換します。
*.nnb モデルのパラメータサイズの縮小¶
2 値化された重みを SIGN データ型で表すことができるように、対応するパラメータのデータ型を設定する必要があります。
学習済みモデルからバッファー設定ファイルをエクスポートします。
$/> nnabla_cli nnb_template bin_class.nnp setting.yaml
出力される setting.yaml は次のようになります。
functions:
...
variables:
...
input: FLOAT32 <-- buffer
conv1/bwn_conv/W: FLOAT32 <-- parameter
conv1/bwn_conv/Wb: FLOAT32 <-- parameter
conv1/bwn_conv/alpha: FLOAT32 <-- parameter
BinaryWeightConvolution_Output: FLOAT32
conv1/bn/beta: FLOAT32
conv1/bn/gamma: FLOAT32
conv1/bn/mean: FLOAT32
conv1/bn/var: FLOAT32
BatchNormalization_Output: FLOAT32
ReLU_Output: FLOAT32
affine/W: FLOAT32
affine/b: FLOAT32
output: FLOAT32
...
名前に基づいて、バッファーとパラメータ型に注釈を付けました。バッファーとパラメータの違いは、パラメータの値は演算実行前に決定されていますが、バッファーの値は演算を実行するまで未定である点です。そのため、量子化ポリシーが異なります。ご存知のように、 conv1/bwn_conv/W は浮動小数点表現のため、使用されませんので無視してください。 conv1/bwn_conv/Wb を “SIGN” 型として識別する必要があり、次のようになります。
functions:
...
variables:
...
input: FLOAT32
conv1/bwn_conv/W: FLOAT32 <-- omit
conv1/bwn_conv/Wb: SIGN <-- identified as SIGN
...
output: FLOAT32
...
テストデータセットに対するトップ1エラーは以下のようになりました。
nnp accuracy: 0.76, nnb accuracy: 0.73
ご覧いただけるように、精度の低下は浮動小数点演算の場合と比較してわずかです。
また、 *.nnb サイズは 830KB から 219KB に縮小されました。
binary_connect_convolution¶
PF.convolution() を PF.binary_connect_convolution() に置き換え、上記と同じ学習を行いました。
以下のような学習速度の劣化と精度劣化が観測されました。
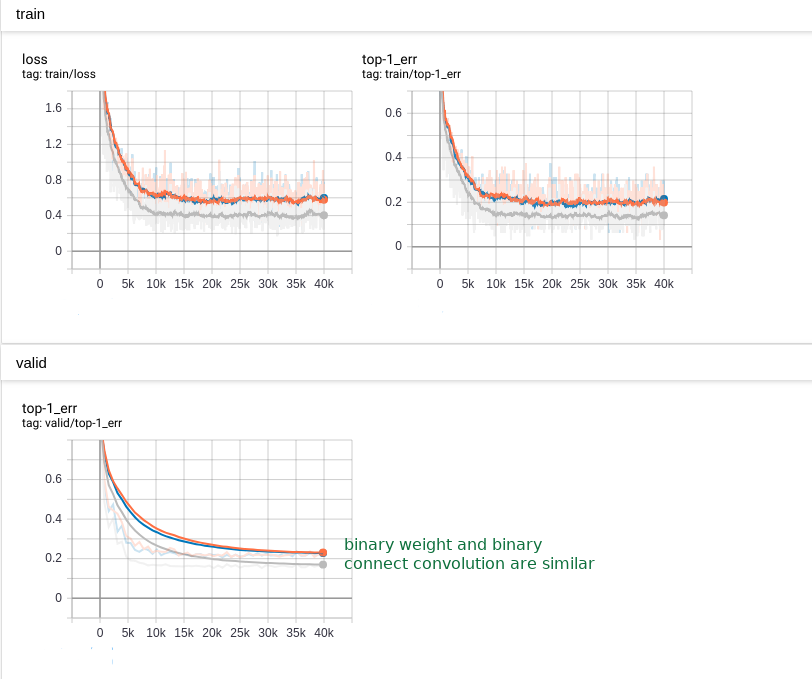
テストデータセットに対するトップ1エラーは以下のようになりました。
nnp accuracy: 0.68, nnb accuracy: 0.71
ご覧のように、精度の低下は見られますが、 nnabla_cli は nnablart よりも低い結果になりました。このテスト結果は、float32による実行結果が二値量子化した場合よりも精度が低いことを示しています。それは問題となります。その理由は、学習プロセスが 2 つのデータパスを介してデータを渡すため、バイナリ重みデータパスの損失が、同時に float32 データパスよりも低くなるためです。
量子化関数¶
binary_weight シリーズ関数と binary_connect シリーズ関数の主な違いは、量子化をする方法(式)です。
binary_weight_convolution または binary_weight_affine については以下の式によって量子化を行います。
binary_connect_convolution または binary_connect_affine については、二値化を行う方法が二つ存在します。1つは、以下の方法です。
もう一つは以下のような方法です。
上式で sigmaは "hard sigmoid" を表し、以下のような式で表されます。
nnabla の実装では、 binary_connect_xxxx() は次の式によって実装されています。
以下のように、それぞれの量子化方法で若干の精度差が見られます。
nnabla で推定される精度 |
nnablart で推定される精度 |
モデルサイズ |
|
|---|---|---|---|
浮動小数点 |
0.81 |
0.81 |
449.5 KB |
binary weight convolution 使用 |
0.76 |
0.75 |
52.1 KB |
binary connect convolution 使用 |
0.68 |
0.71 |
47.3 KB |
モデルサイズは既に約10倍小さくなっており、劇的に削減されています。
フットプリントをさらに削減¶
最大限の精度を維持し、フットプリントを可能な限り減らす為に、前述の 2 番目の方法を試してみましょう。この方法は、学習済みのネットワークに対して、有効に働く傾向があります。量子化プロセスは学習後に行われます。ここでは、浮動小数点演算で学習済みのモデルを利用して、実験を行います。
上述のベンチマークネットワークの解析から、バッファーサイズがフットプリントの大半を占めることがすでに分かっています。次の図では、円はVariableのバッファーを表し、長方形は関数を表します。Function 1 を実行する際には、buffer1, 2, 3に相当するメモリ領域が確保されます。Function 1 の実行後、Function 2が実行される際には、buffer 1, 2は解放されています。このとき、もしbuffer1のメモリサイズが十分大きくFunction 2の出力を保持できる際には、buffer 1のメモリ領域が再利用されます。
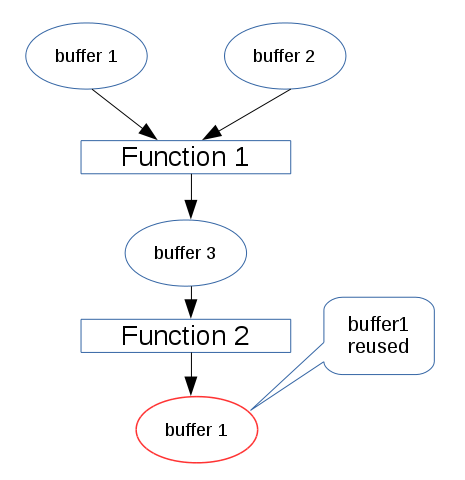
このバッファー再利用ポリシーは、 *.nnp から *.nnb への変換中に実装されています。各関数の占有メモリの最大値は、このネットワークによって推論を行う際の最大フットプリントメモリ量に相当します。このサイズを削減するために、Variableのバッファーに量子化データ型を使用する場合があります。前の setting.yaml と同様に、次のバッファー型が変更された場合、 *.nnp から *.nnb に変換すると、この新しいバッファー型の定義に基づいてバッファーサイズが計算されます。
functions:
...
variables:
...
input: FLOAT32 ==> input: FIXED8
conv1/bwn_conv/W: FIXED8
conv1/bwn_conv/Wb: FIXED8
...
output: FLOAT32
...
この量子化プロセスよって発生する量子化ノイズによって、精度が明らかに劣化する場合があることが広く知られています。最適な量子化ステップサイズを選択する方法については、 [2] を参照してください。
固定小数点の位置の決定¶
*.nnp から *.nnb への変換では、それぞれのパラメータの値のヒストグラム(または分布)がその時点でわかっている為、パラメータ型の量子化時に固定小数点位置を自動的に決定できます。Variableのバッファーの固定小数点位置に起因した歪みを最小限に抑えるために、variableのバッファーについてもその値のヒストグラム(または分布)に応じて固定小数点位置を決定する必要があります。しかしながら、学習中に統計をとっても、各Variableのバッファーの分布を正確に知ることは困難です。今後のテストデータセットの分布は、現在の既知のデータセットと同じであると仮定し、現在の既知のデータセットに基づいて固定小数点の決定を行います。
固定小数点位置を手動で調整することは、芸術のような作業です。ここではいくつかの経験的な知見を共有しましたが、より賢く自動化された方法が必要となるでしょう。
次の図のように、既知の小さなデータセットで変数バッファーの分布を収集しました。(すべての変数分布がここにリストされているわけではありません。)
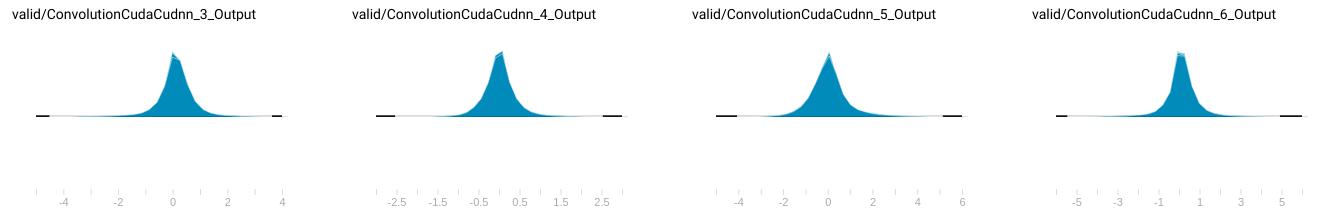
バッファー値の分布¶
もちろん、固定小数点位置を決定する最も簡単な方法は、変数バッファーで発生する最小値と最大値をを出力して決定することですが、その場合には、めったに出現しないような外れ値を表現するために、値の範囲を広げることになる可能性もあり、結果的に小数部の精度を劣化させてしまうことになります。
この分布に従って、 FP_POS を次のように計算しました。(new_setting.yaml)
variables:
...
Convolution_3_Output: FIXED16_12 <-- Change data type according to value distribution
conv2/conv2/bn/beta: FLOAT32
conv2/conv2/bn/gamma: FLOAT32
conv2/conv2/bn/mean: FLOAT32
conv2/conv2/bn/var: FLOAT32
BatchNormalization_3_Output: FIXED16_12
ReLU_3_Output: FIXED16_12
conv2/conv3/conv/W: FIXED8
Convolution_4_Output: FIXED16_12
conv2/conv3/bn/beta: FLOAT32
conv2/conv3/bn/gamma: FLOAT32
conv2/conv3/bn/mean: FLOAT32
conv2/conv3/bn/var: FLOAT32
BatchNormalization_4_Output: FIXED8_4
Add2_Output: FIXED8_4
ReLU_4_Output: FIXED8_4
...
new_setting.yaml を変更した後、以下のコマンドによって、 *.nnp から *.nnb へ変換を行います。
$> nnabla_cli convert -b 1 -d nnb_3 models/bin_class_float.nnp models/bin_class_f_fq.nnb -s setting/setting_f_fq.yaml
-d nnb_3 は、メモリ節約ポリシーを有効にするために必要です。 チューニングすることで、最先端の結果を得ました。
nnp accuracy: 0.81, nnb accuracy: 0.79
まとめ¶
Variableのバッファーを量子化することによって、以下の表に示すように、フットプリントは1.2Mから495.2Kとなり、明らかに削減され、かつ精度はほとんどそのままに維持されています。
モデルサイズ |
フットプリント |
精度 |
|
|---|---|---|---|
浮動小数点モデル |
449.5 KB |
1.2 M |
0.81 |
量子化されたパラメータ |
126.0 KB |
1.0 M |
0.81 |
量子化されたパラメータとバッファー |
126.0 KB |
495.2 KB |
0.79 |
これら 2 つの方法を比較すると、現在のnnablaの実装では、2番目の方法の方が良い結果となっています。その理由は、現在のnnablaではSIGN型のパラメータをサポートしていないので、重みの二値化によるメモリ削減の恩恵をフルに受けられないまま精度劣化のみが起きているからであると考えられます。また、今後の改善として、二値化処理を行う関数については、浮動小数点の重みパラメータを *.nnb から削除する必要があるでしょう。
注意: 現在、すべての実験は分類問題に焦点を当てていて、softmax は量子化許容値の影響を減らします。回帰問題に対するテストはまだ行っておらず、どの程度精度が劣化するかは不明です。